Регрессии
сентябрь 2021г.
1 Задание 1 – проверка простых гипотез
См. Работающий пример в Лекции 3
Используя образец из лекции, постройте регрессионную модель, которая проверяла бы те же гипотезы, что и отдельные тесты на прошлом семинаре:
1.1 Гипотезы
Важность ценности веселья impfun у жителей Швейцарии (код CH в переменной cntry) связана с:
- полом
gndr(мужчины в большей степени склонны разделять ценности веселья), - возрастом
agea(более молодые люди в большей степени привержены ценности веселья); - и образованием
eduyrs(более образованные жители Швейцарии сильнее склонны разделять ценность веселья, чем менее образованные); - регион, в котором проживает респондент
region. Выясните, какие регионы значимо различаются.
Дополнительно: возраст имеет более сильный эффект на ценность веселья, чем образование.
Проверьте эти гипотезы на данных 8 раунда ESS.
# считываем данные
library("foreign")
d <- read.spss("data/ESS8e02.sav",
use.value.labels = F,
use.missings = T,
to.data.frame = T
)
# отбираем швейцарцев
CH <- d[d$cntry == "CH",]
# меняем кодировку на обратную
CH$impfun.rec <- 7 - CH$impfun
# строим регрессионную модель
fit1 <- lm(impfun.rec ~ gndr + agea + eduyrs + region, CH)
# делаем таблицу регрессионных коэффициентов
library("texreg")
library("htmltools")
htab1 <- htmlreg(fit1)
browsable(HTML(htab1))| Model 1 | |
|---|---|
| (Intercept) | 5.63*** |
| (0.20) | |
| gndr | -0.12 |
| (0.06) | |
| agea | -0.01*** |
| (0.00) | |
| eduyrs | -0.03** |
| (0.01) | |
| regionCH02 | -0.34*** |
| (0.10) | |
| regionCH03 | -0.34** |
| (0.11) | |
| regionCH04 | -0.45*** |
| (0.11) | |
| regionCH05 | -0.13 |
| (0.11) | |
| regionCH06 | -0.30* |
| (0.13) | |
| regionCH07 | -0.78*** |
| (0.18) | |
| R2 | 0.07 |
| Adj. R2 | 0.06 |
| Num. obs. | 1508 |
| ***p < 0.001; **p < 0.01; *p < 0.05 | |
2 Задание 2 – квадратические связи
- Стандартизируйте все интервальные переменные в модели и заново вычислите коэффициенты.
- Проверьте криволинейный эффект возраста и количества лет образования, дополнив модель их квадратами. Квадраты можно вводить непосредственно в формулу в виде
I(predictor).
Обертка
I()необходима для правильного считывания формулы, функцияI()изолирует выражение и позволяет совершать разные преобразования внутри формулы. Это необходимо для последующего построения графиков.
- Если какой-то из квадратических эффектов обнаружится, постройте график, демонстрирующий эту связь, используя функцию
plot_model()и аргументtype = "eff".
# вычисляем стандартизованные коэффициенты
fit2 <- lm(scale(impfun.rec) ~
gndr + scale(agea) + scale(eduyrs) + region,
CH
)
# смотрим на таблицу коэффициентов
htab2 <- htmlreg(fit2)
browsable(HTML(htab2))| Model 1 | |
|---|---|
| (Intercept) | 0.37*** |
| (0.10) | |
| gndr | -0.10 |
| (0.05) | |
| scale(agea) | -0.21*** |
| (0.03) | |
| scale(eduyrs) | -0.07** |
| (0.03) | |
| regionCH02 | -0.27*** |
| (0.08) | |
| regionCH03 | -0.27** |
| (0.09) | |
| regionCH04 | -0.35*** |
| (0.09) | |
| regionCH05 | -0.10 |
| (0.09) | |
| regionCH06 | -0.24* |
| (0.10) | |
| regionCH07 | -0.61*** |
| (0.14) | |
| R2 | 0.07 |
| Adj. R2 | 0.06 |
| Num. obs. | 1508 |
| ***p < 0.001; **p < 0.01; *p < 0.05 | |
# добавили квадратические члены
fit3 <- lm(scale(impfun.rec) ~
gndr + scale(agea) + I(agea^2) +
scale(eduyrs) + I(eduyrs^2) +
region,
CH
)
# смотрим на таблицу коэффициентов
htab3 <- htmlreg(fit3)
browsable(HTML(htab3))| Model 1 | |
|---|---|
| (Intercept) | 0.14 |
| (0.30) | |
| gndr | -0.10 |
| (0.05) | |
| scale(agea) | -0.50*** |
| (0.13) | |
| agea^2 | 0.00* |
| (0.00) | |
| scale(eduyrs) | 0.07 |
| (0.14) | |
| eduyrs^2 | -0.00 |
| (0.00) | |
| regionCH02 | -0.27*** |
| (0.08) | |
| regionCH03 | -0.27** |
| (0.09) | |
| regionCH04 | -0.36*** |
| (0.09) | |
| regionCH05 | -0.10 |
| (0.09) | |
| regionCH06 | -0.23* |
| (0.10) | |
| regionCH07 | -0.62*** |
| (0.14) | |
| R2 | 0.07 |
| Adj. R2 | 0.07 |
| Num. obs. | 1508 |
| ***p < 0.001; **p < 0.01; *p < 0.05 | |
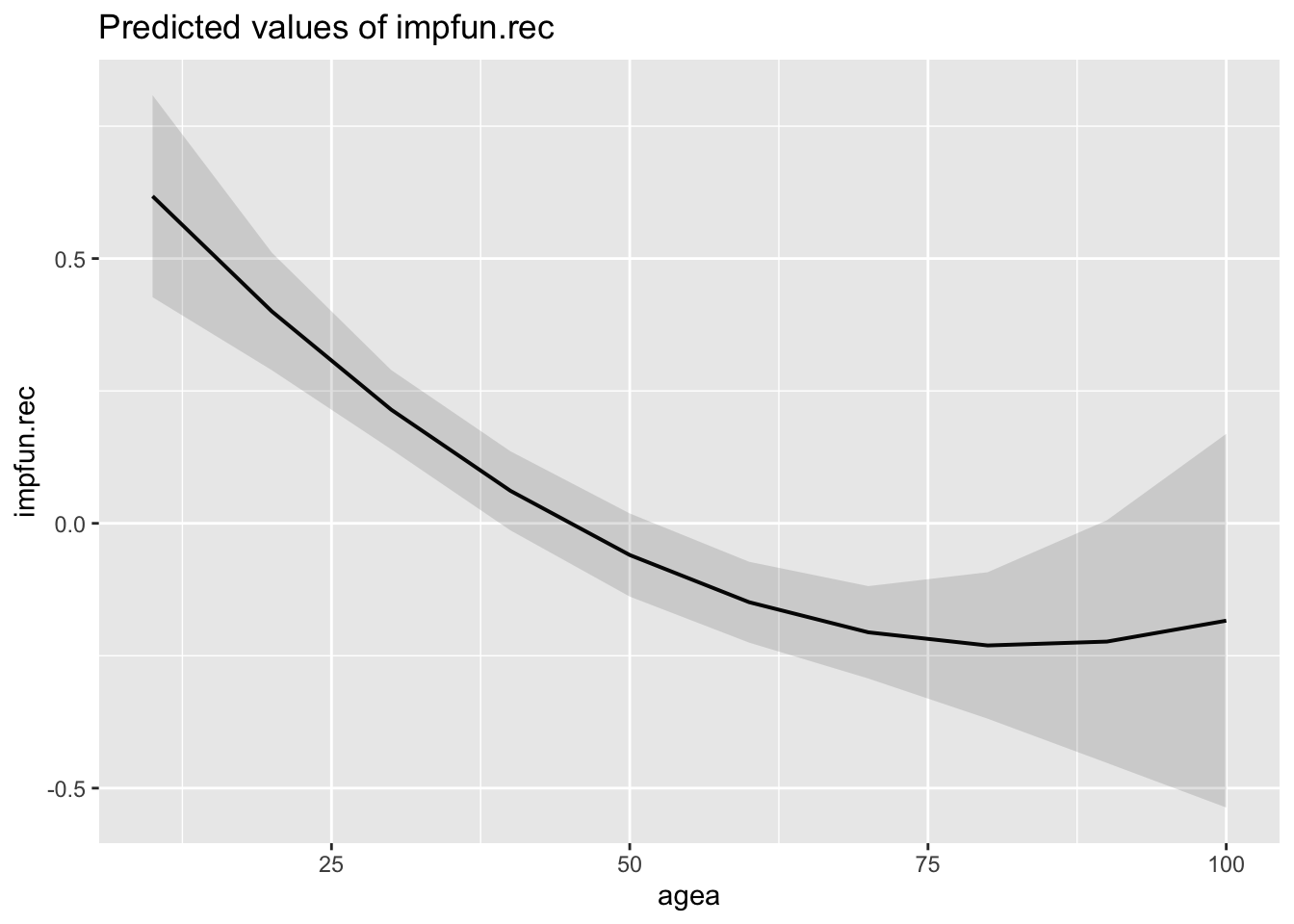
3 Задание 3 – диагностика
Проведите диагностику моделей
- высок ли R2?
- мультиколлинеарность (
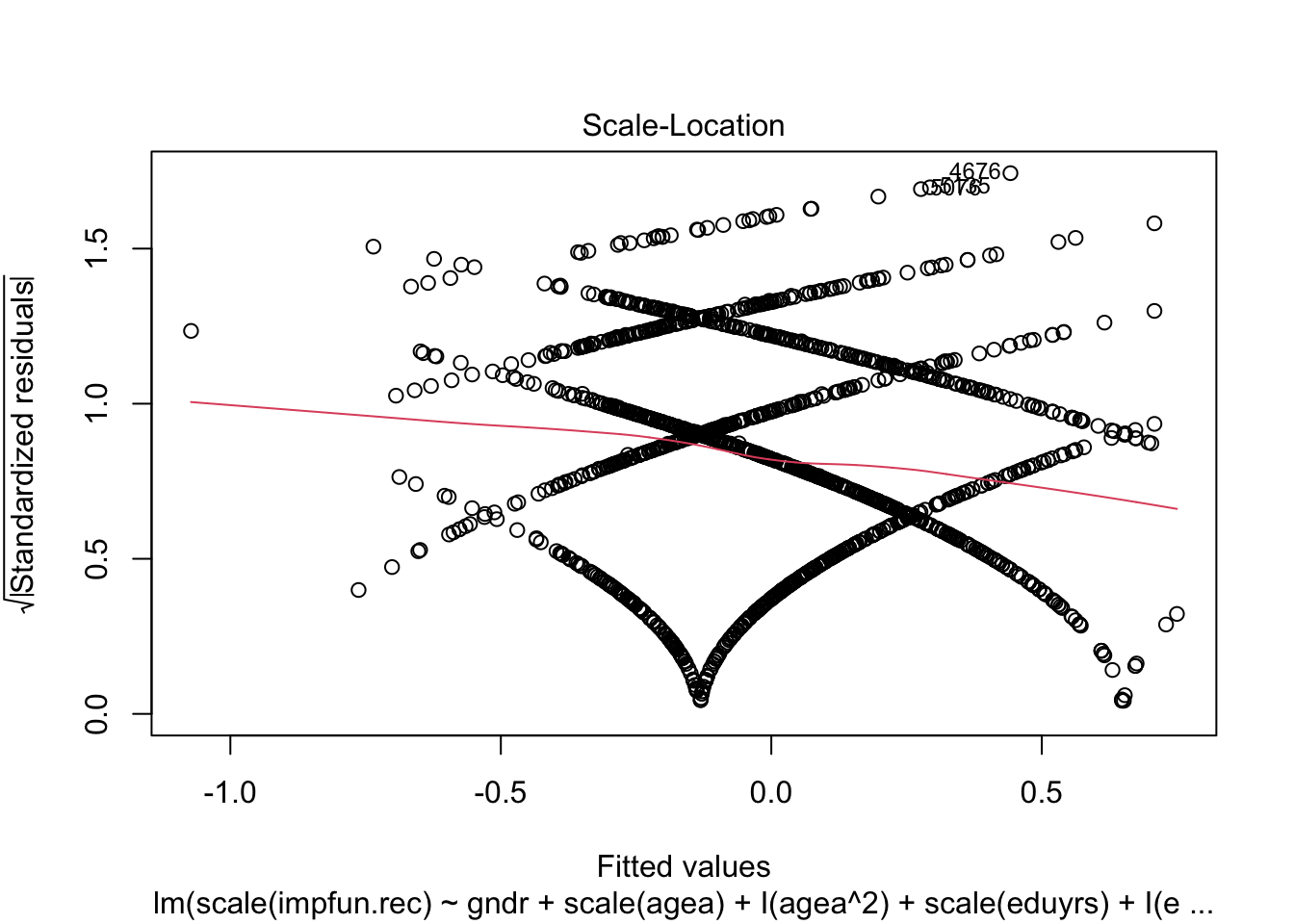
car::vif(), проблема если >5 или >10); - гомоскедастичность
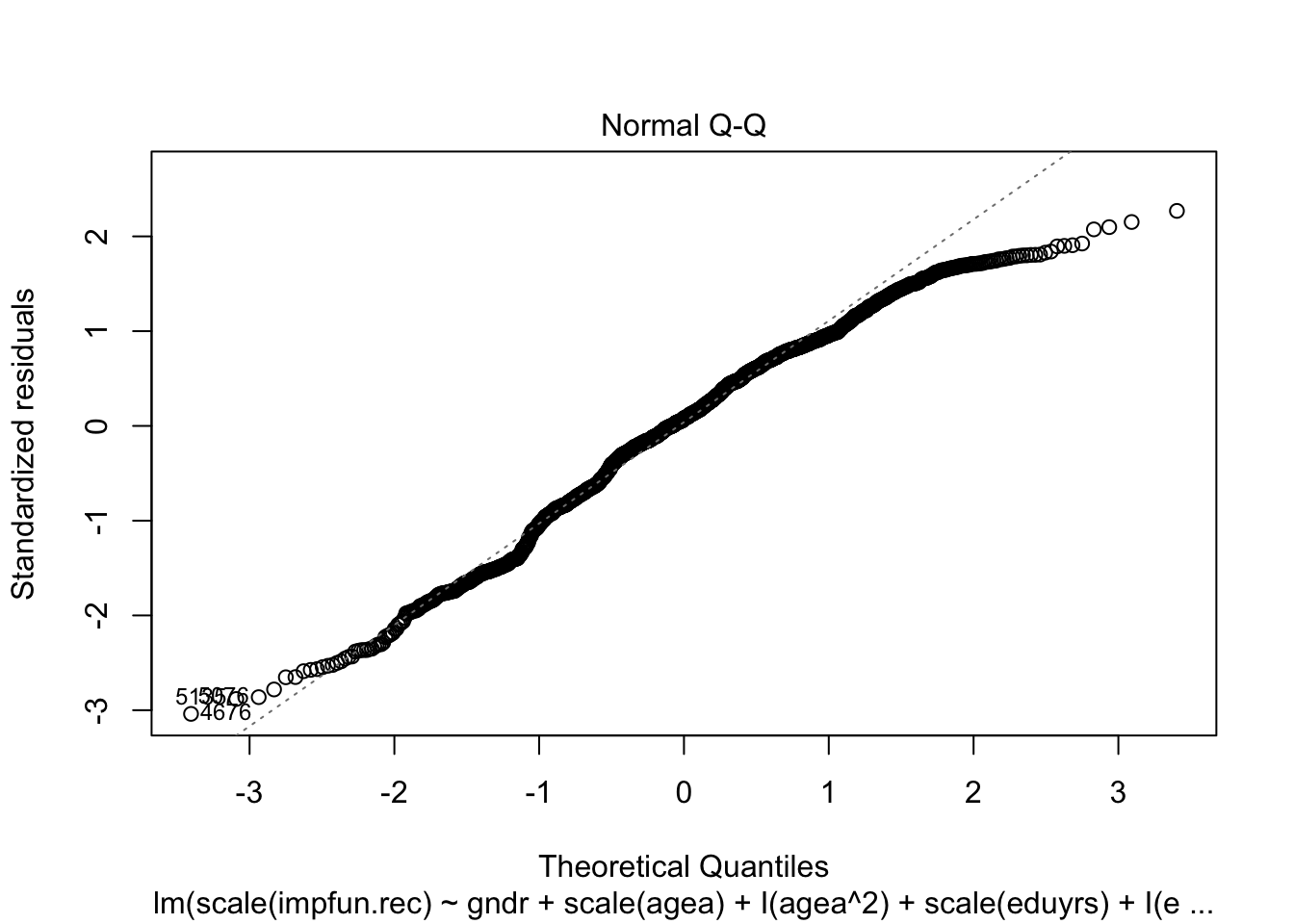
lmtest::bptest()тест Бреуша-Пэгэна, тест непостоянной дисперсии остатков (значимый указывает на наличие гетероскедастичности); - нормальность распределения остатков
shapiro.test() plot()- диагностические графики.
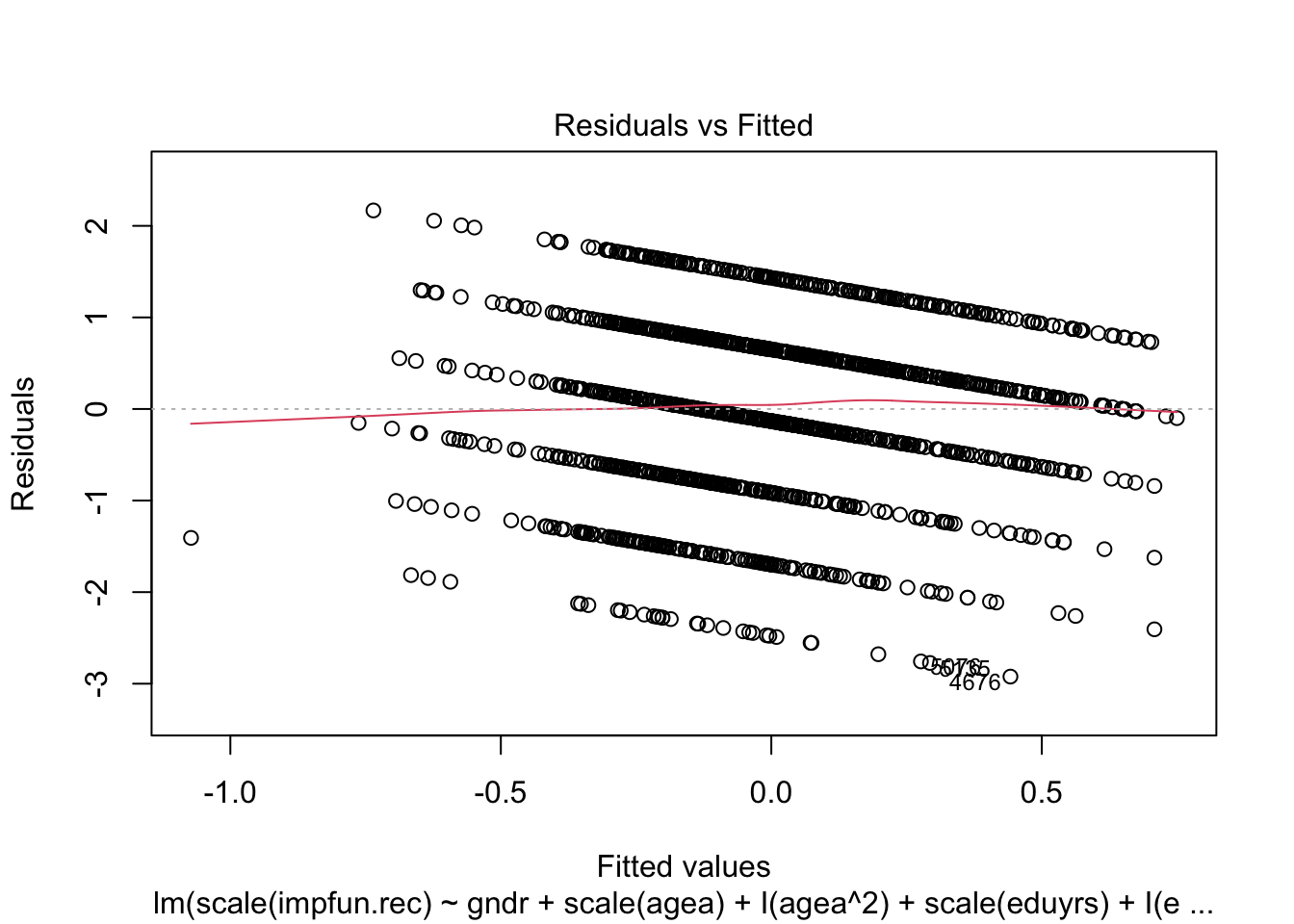
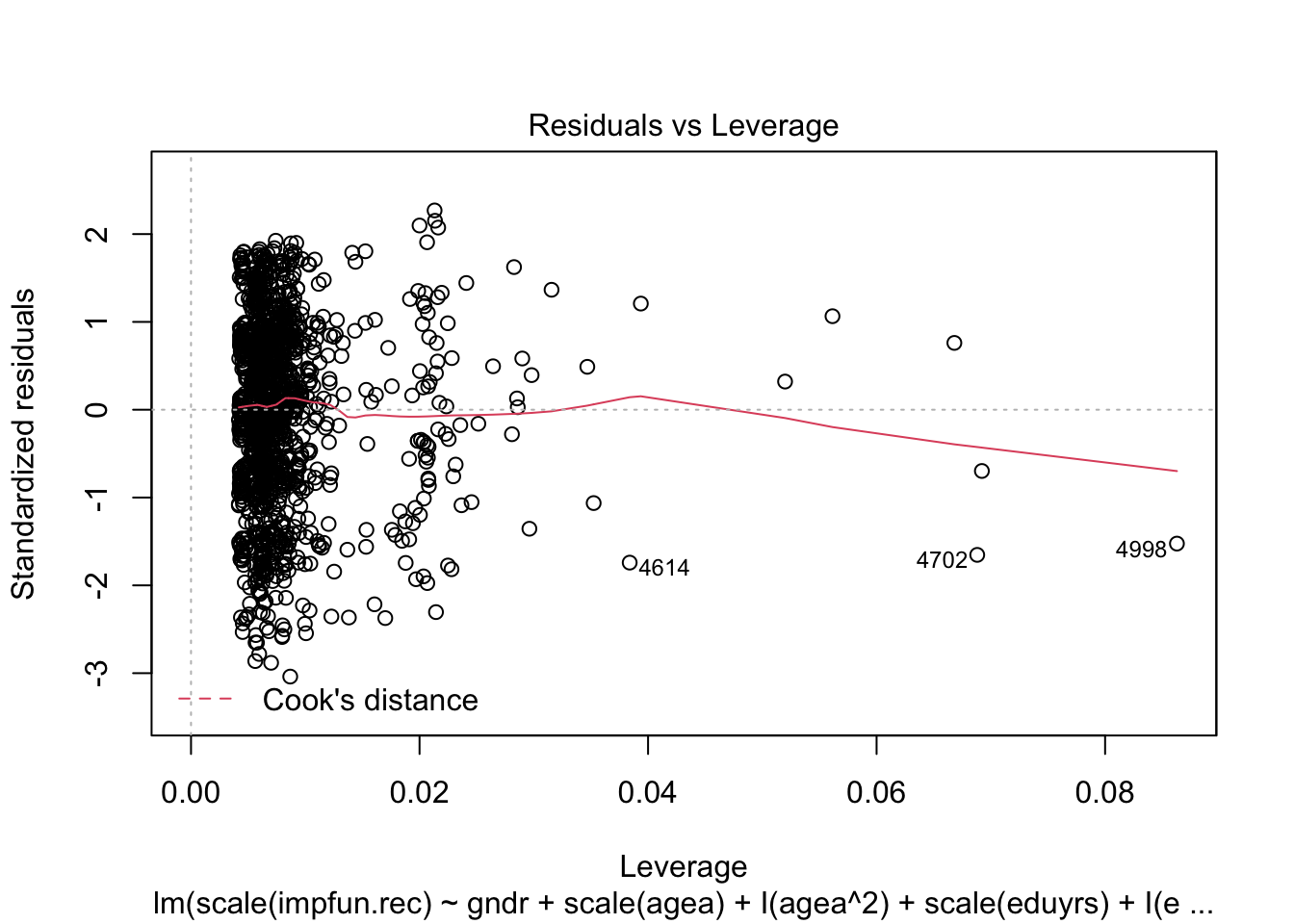
> GVIF Df GVIF^(1/(2*Df))
> gndr 1.006127 1 1.003059
> scale(agea) 27.258541 1 5.220971
> I(agea^2) 27.461828 1 5.240403
> scale(eduyrs) 30.336066 1 5.507819
> I(eduyrs^2) 30.277220 1 5.502474
> region 1.084653 6 1.006795>
> studentized Breusch-Pagan test
>
> data: fit3
> BP = 28.967, df = 11, p-value = 0.002297> Non-constant Variance Score Test
> Variance formula: ~ fitted.values
> Chisquare = 20.13085, Df = 1, p = 7.2321e-06>
> Shapiro-Wilk normality test
>
> data: residuals(fit3)
> W = 0.98215, p-value = 9.889e-13# ОПЦИОНАЛЬНО: зависимость остатков от предикторов - проверка на эндогенность - опциональная
summary(lm(residuals(fit3) ~
fit3$model$gndr +
fit3$model$`scale(agea)` + fit3$model$`I(agea^2)` +
fit3$model$`scale(eduyrs)` + fit3$model$`I(eduyrs^2)` +
fit3$model$region))>
> Call:
> lm(formula = residuals(fit3) ~ fit3$model$gndr + fit3$model$`scale(agea)` +
> fit3$model$`I(agea^2)` + fit3$model$`scale(eduyrs)` + fit3$model$`I(eduyrs^2)` +
> fit3$model$region)
>
> Residuals:
> Min 1Q Median 3Q Max
> -2.92234 -0.65386 0.07653 0.73301 2.16811
>
> Coefficients:
> Estimate Std. Error t value Pr(>|t|)
> (Intercept) 7.277e-17 3.040e-01 0 1
> fit3$model$gndr 4.888e-17 4.993e-02 0 1
> fit3$model$`scale(agea)` 1.463e-16 1.301e-01 0 1
> fit3$model$`I(agea^2)` -7.914e-20 7.029e-05 0 1
> fit3$model$`scale(eduyrs)` -1.864e-16 1.374e-01 0 1
> fit3$model$`I(eduyrs^2)` 1.208e-18 1.493e-03 0 1
> fit3$model$regionCH02 3.980e-17 7.886e-02 0 1
> fit3$model$regionCH03 2.188e-16 8.995e-02 0 1
> fit3$model$regionCH04 1.853e-16 8.734e-02 0 1
> fit3$model$regionCH05 -1.122e-16 8.909e-02 0 1
> fit3$model$regionCH06 -1.432e-17 9.871e-02 0 1
> fit3$model$regionCH07 -1.484e-16 1.435e-01 0 1
>
> Residual standard error: 0.9659 on 1496 degrees of freedom
> Multiple R-squared: 4.473e-32, Adjusted R-squared: -0.007353
> F-statistic: 6.083e-30 on 11 and 1496 DF, p-value: 1# ОПЦИОНАЛЬНО: корреляция между остатками и предсказанными значениями y - проверка на гетероскедастичность
cor(
sqrt(abs(residuals(fit3))),
predict(fit3)
)> [1] -0.1348646>
> Shapiro-Wilk normality test
>
> data: CH$impfun.rec
> W = 0.91424, p-value < 2.2e-16# ОПЦИОНАЛЬНО: Еше один тест на нормальность распределения остатков
ks.test(residuals(fit3), "pnorm", 0, sd(residuals(fit3))) # ties == repeated values>
> One-sample Kolmogorov-Smirnov test
>
> data: residuals(fit3)
> D = 0.04793, p-value = 0.001959
> alternative hypothesis: two-sided4 Задание 4 – интеракции
5 Взаимодействие предикторов – модерация
http://apps.maksimrudnev.com/learn/paradox.Rmd
- Добавьте в модель взаимодействие возраста и гендера. Постройте график интеракций и проитерпретируйте коэффициенты.
# Добавляем интеракцию между переменными используя символ звездочка *
fit4 <- lm(scale(impfun.rec) ~
gndr * scale(agea) + I(agea^2) +
scale(eduyrs) + I(eduyrs^2) +
region,
CH
)
# смотрим на результаты
htab4 <- htmlreg(fit4)
browsable(HTML(htab4))| Model 1 | |
|---|---|
| (Intercept) | 0.10 |
| (0.30) | |
| gndr | -0.10 |
| (0.05) | |
| scale(agea) | -0.29* |
| (0.15) | |
| agea^2 | 0.00* |
| (0.00) | |
| scale(eduyrs) | 0.05 |
| (0.14) | |
| eduyrs^2 | -0.00 |
| (0.00) | |
| regionCH02 | -0.27*** |
| (0.08) | |
| regionCH03 | -0.27** |
| (0.09) | |
| regionCH04 | -0.36*** |
| (0.09) | |
| regionCH05 | -0.10 |
| (0.09) | |
| regionCH06 | -0.23* |
| (0.10) | |
| regionCH07 | -0.61*** |
| (0.14) | |
| gndr:scale(agea) | -0.16** |
| (0.05) | |
| R2 | 0.08 |
| Adj. R2 | 0.07 |
| Num. obs. | 1508 |
| ***p < 0.001; **p < 0.01; *p < 0.05 | |
# обязательно визуализируем для более точной интерпретации
plot_model(fit4, type = "eff", terms = c("agea", "gndr"))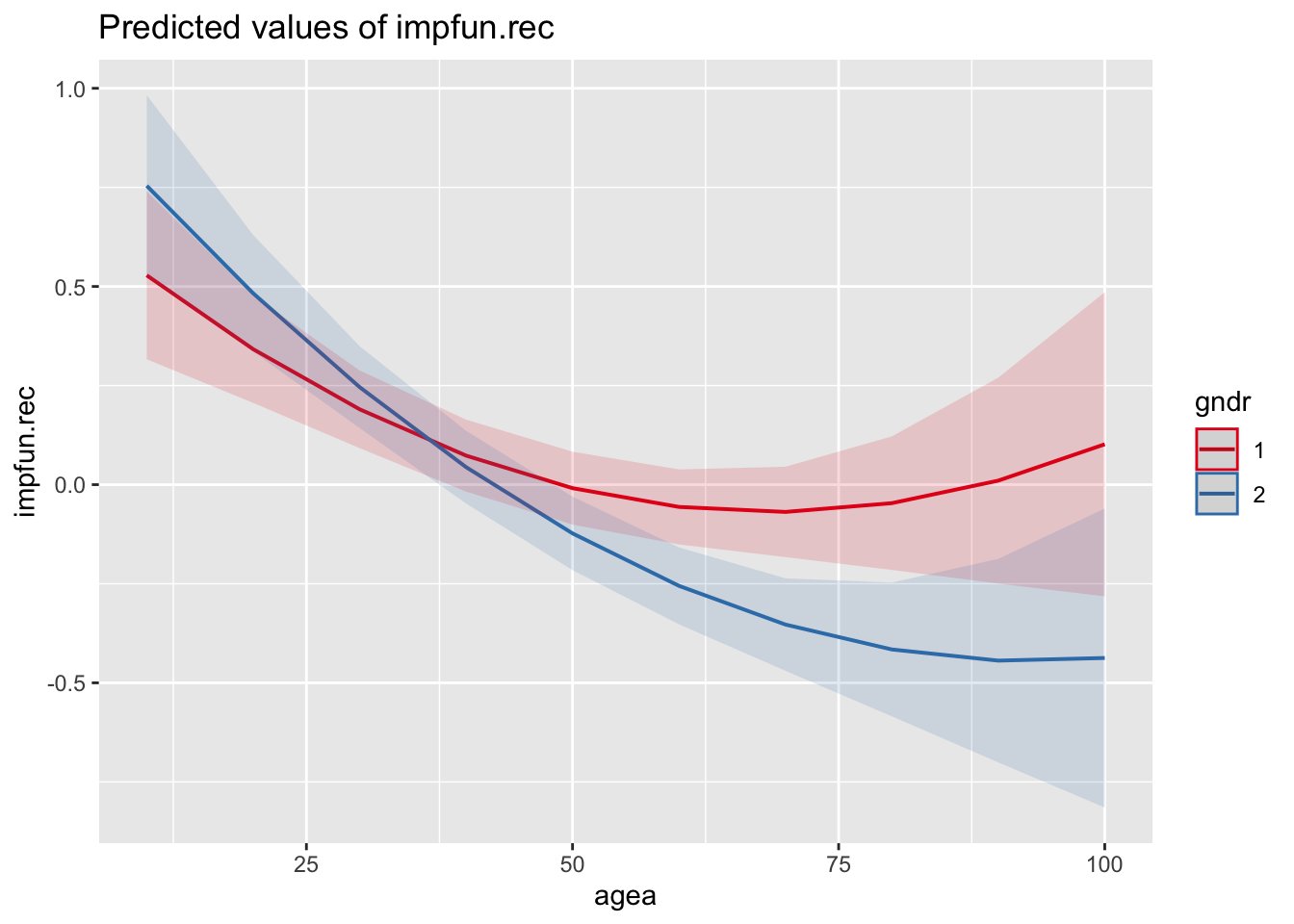
# сводим все модели в одну таблицу
htab.final <- htmlreg(list(fit1, fit2, fit3, fit4))
browsable(HTML(htab.final))| Model 1 | Model 2 | Model 3 | Model 4 | |
|---|---|---|---|---|
| (Intercept) | 5.63*** | 0.37*** | 0.14 | 0.10 |
| (0.20) | (0.10) | (0.30) | (0.30) | |
| gndr | -0.12 | -0.10 | -0.10 | -0.10 |
| (0.06) | (0.05) | (0.05) | (0.05) | |
| agea | -0.01*** | |||
| (0.00) | ||||
| eduyrs | -0.03** | |||
| (0.01) | ||||
| regionCH02 | -0.34*** | -0.27*** | -0.27*** | -0.27*** |
| (0.10) | (0.08) | (0.08) | (0.08) | |
| regionCH03 | -0.34** | -0.27** | -0.27** | -0.27** |
| (0.11) | (0.09) | (0.09) | (0.09) | |
| regionCH04 | -0.45*** | -0.35*** | -0.36*** | -0.36*** |
| (0.11) | (0.09) | (0.09) | (0.09) | |
| regionCH05 | -0.13 | -0.10 | -0.10 | -0.10 |
| (0.11) | (0.09) | (0.09) | (0.09) | |
| regionCH06 | -0.30* | -0.24* | -0.23* | -0.23* |
| (0.13) | (0.10) | (0.10) | (0.10) | |
| regionCH07 | -0.78*** | -0.61*** | -0.62*** | -0.61*** |
| (0.18) | (0.14) | (0.14) | (0.14) | |
| scale(agea) | -0.21*** | -0.50*** | -0.29* | |
| (0.03) | (0.13) | (0.15) | ||
| scale(eduyrs) | -0.07** | 0.07 | 0.05 | |
| (0.03) | (0.14) | (0.14) | ||
| agea^2 | 0.00* | 0.00* | ||
| (0.00) | (0.00) | |||
| eduyrs^2 | -0.00 | -0.00 | ||
| (0.00) | (0.00) | |||
| gndr:scale(agea) | -0.16** | |||
| (0.05) | ||||
| R2 | 0.07 | 0.07 | 0.07 | 0.08 |
| Adj. R2 | 0.06 | 0.06 | 0.07 | 0.07 |
| Num. obs. | 1508 | 1508 | 1508 | 1508 |
| ***p < 0.001; **p < 0.01; *p < 0.05 | ||||
6 👉🏻 Большое задание
Вычислите переменную – общее количество выпиваемого россиянами в неделю, используя две переменные:
alcwknd- количество выпитого алкоголя на выходных;alcwkdy- количество выпитого алкоголя в рабочие дни.
Удалите из массива тех, кто не пил на этой неделе.
Попробуйте ответить на следующие вопросы:
- Сколько всего в неделю выпивают российские мужчины и женщины (
gndr)? - Коррелирует ли употребление алкоголя с возрастом (
agea) и образованием (eduyrs)? - Влияют ли возраст, гендер, образование, наличие работы (
icpdwrk) и партнера (icpart1), а также частота встреч с друзьямиsclmeetи тип населенного пунктаdomicilна употребление алкоголя? Постройте регрессионные модели пошагово вводя каждую из независимых переменных. Меняются ли коэффициенты с добавлением предикторов? Почему?
# Считываем данные
library(foreign)
ess7 <- read.spss("data/Datafile_ESS7.sav", use.value.labels = F, to.data.frame=T)
# Отбираем россиян
ess7.RU <- ess7[ess7$cntry == "RU",]
# Делаем перекодировку для удобства
ess7.RU$gender <- factor(ess7.RU$gndr, labels = c("Male", "Female") )
ess7.RU$domicil <- factor(ess7.RU$domicil)
# Вычисляем зависимую переменную
ess7.RU$alcwkdy[ess7.RU$alcwkdy==5555] <- 0
ess7.RU$alcwknd[ess7.RU$alcwknd==5555] <- 0
ess7.RU$alc <- ess7.RU$alcwkdy + ess7.RU$alcwknd
ess7.RU$alc[ess7.RU$alc==0] <- NA
# Смотрим на распределение зависимой переменной
hist(ess7.RU$alc)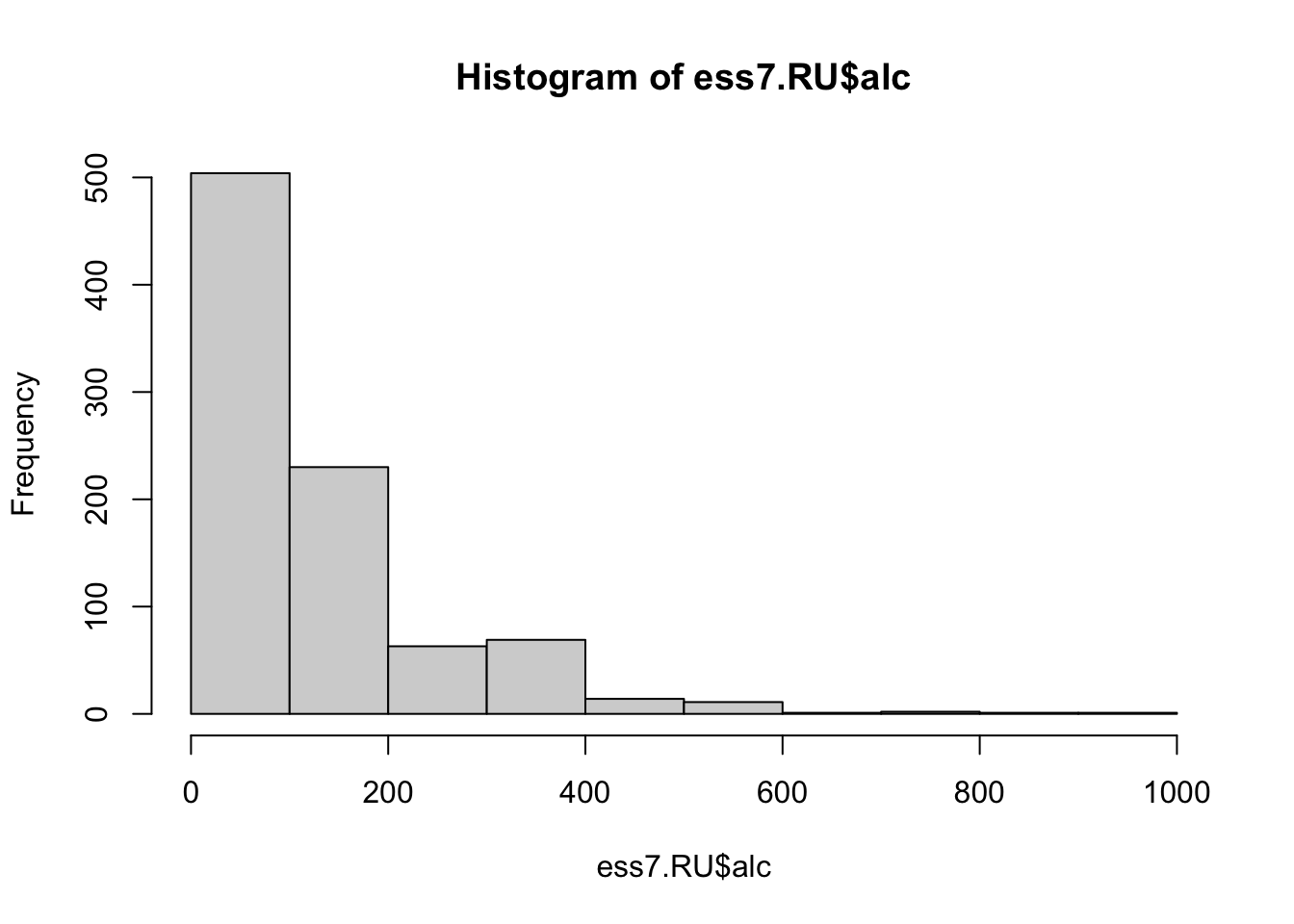
# Пошагово вводим предикторы
fit1 <- lm(alc ~
agea ,
data= ess7.RU
)
fit2 <- lm(alc ~
agea + gender ,
data= ess7.RU
)
fit3 <- lm(alc ~
agea + gender + eduyrs,
data= ess7.RU
)
fit4 <- lm(alc ~
agea + gender + eduyrs +
sclmeet ,
data= ess7.RU
)
fit5 <- lm(alc ~
agea + gender + eduyrs +
sclmeet + icpart1 + icpdwrk + domicil,
data= ess7.RU
)
# Строим общую таблицу
regtab <- htmlreg(list(fit1, fit2, fit3, fit4, fit5))
browsable(HTML(regtab))| Model 1 | Model 2 | Model 3 | Model 4 | Model 5 | |
|---|---|---|---|---|---|
| (Intercept) | 101.26*** | 125.18*** | 180.34*** | 182.46*** | 162.80*** |
| (12.66) | (12.62) | (25.87) | (29.51) | (34.45) | |
| agea | 0.77** | 0.92*** | 0.78** | 0.77** | 0.88** |
| (0.27) | (0.27) | (0.27) | (0.28) | (0.29) | |
| genderFemale | -63.46*** | -60.85*** | -59.76*** | -62.23*** | |
| (8.09) | (8.14) | (8.25) | (8.52) | ||
| eduyrs | -3.87* | -4.15** | -3.81* | ||
| (1.58) | (1.60) | (1.64) | |||
| sclmeet | 0.27 | -0.24 | |||
| (2.62) | (2.65) | ||||
| icpart1 | 8.06 | ||||
| (8.56) | |||||
| icpdwrk | -4.89 | ||||
| (9.68) | |||||
| domicil2 | 69.99** | ||||
| (24.54) | |||||
| domicil3 | 17.13 | ||||
| (9.49) | |||||
| domicil4 | 3.72 | ||||
| (10.84) | |||||
| domicil5 | 5.86 | ||||
| (60.55) | |||||
| R2 | 0.01 | 0.07 | 0.08 | 0.08 | 0.09 |
| Adj. R2 | 0.01 | 0.07 | 0.08 | 0.07 | 0.08 |
| Num. obs. | 896 | 896 | 896 | 883 | 883 |
| ***p < 0.001; **p < 0.01; *p < 0.05 | |||||
- Проверьте регрессионную модель на гетероскедастичность и мультиколлинеарность.
>
> Call:
> lm(formula = alc ~ agea + gender + eduyrs + sclmeet + icpart1 +
> icpdwrk + domicil, data = ess7.RU)
>
> Residuals:
> Min 1Q Median 3Q Max
> -198.47 -73.82 -33.06 37.87 737.50
>
> Coefficients:
> Estimate Std. Error t value Pr(>|t|)
> (Intercept) 162.8038 34.4523 4.725 2.68e-06 ***
> agea 0.8812 0.2933 3.005 0.00273 **
> genderFemale -62.2349 8.5215 -7.303 6.33e-13 ***
> eduyrs -3.8147 1.6424 -2.323 0.02043 *
> sclmeet -0.2398 2.6504 -0.090 0.92794
> icpart1 8.0582 8.5625 0.941 0.34692
> icpdwrk -4.8857 9.6805 -0.505 0.61390
> domicil2 69.9915 24.5374 2.852 0.00444 **
> domicil3 17.1305 9.4897 1.805 0.07139 .
> domicil4 3.7222 10.8393 0.343 0.73138
> domicil5 5.8584 60.5542 0.097 0.92295
> ---
> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
>
> Residual standard error: 120.1 on 872 degrees of freedom
> (1562 observations deleted due to missingness)
> Multiple R-squared: 0.09041, Adjusted R-squared: 0.07998
> F-statistic: 8.668 on 10 and 872 DF, p-value: 1.288e-13>
> studentized Breusch-Pagan test
>
> data: fit5
> BP = 33.089, df = 10, p-value = 0.0002631> Non-constant Variance Score Test
> Variance formula: ~ fitted.values
> Chisquare = 107.8896, Df = 1, p = < 2.22e-16> GVIF Df GVIF^(1/(2*Df))
> agea 1.217528 1 1.103416
> gender 1.109469 1 1.053314
> eduyrs 1.125247 1 1.060777
> sclmeet 1.072082 1 1.035414
> icpart1 1.119981 1 1.058291
> icpdwrk 1.207232 1 1.098741
> domicil 1.062740 4 1.007635- Что влияет сильнее – возраст или образование? (Сравните стандартизованные коэффициенты).
# Стандартизируем интервальные переменные в модели
fit6 <- lm(scale(alc) ~
scale(agea) + gender + scale(eduyrs) +
scale(sclmeet) + icpart1 + icpdwrk + domicil,
data= ess7.RU
)
summary(fit6)>
> Call:
> lm(formula = scale(alc) ~ scale(agea) + gender + scale(eduyrs) +
> scale(sclmeet) + icpart1 + icpdwrk + domicil, data = ess7.RU)
>
> Residuals:
> Min 1Q Median 3Q Max
> -1.5857 -0.5898 -0.2642 0.3026 5.8923
>
> Coefficients:
> Estimate Std. Error t value Pr(>|t|)
> (Intercept) 0.15450 0.14166 1.091 0.27573
> scale(agea) 0.12539 0.04173 3.005 0.00273 **
> genderFemale -0.49723 0.06808 -7.303 6.33e-13 ***
> scale(eduyrs) -0.08686 0.03740 -2.323 0.02043 *
> scale(sclmeet) -0.00318 0.03515 -0.090 0.92794
> icpart1 0.06438 0.06841 0.941 0.34692
> icpdwrk -0.03904 0.07734 -0.505 0.61390
> domicil2 0.55921 0.19604 2.852 0.00444 **
> domicil3 0.13687 0.07582 1.805 0.07139 .
> domicil4 0.02974 0.08660 0.343 0.73138
> domicil5 0.04681 0.48380 0.097 0.92295
> ---
> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
>
> Residual standard error: 0.9597 on 872 degrees of freedom
> (1562 observations deleted due to missingness)
> Multiple R-squared: 0.09041, Adjusted R-squared: 0.07998
> F-statistic: 8.668 on 10 and 872 DF, p-value: 1.288e-13- Введите интеракцию (взаимодействие) между гендером и возрастом и попробуйте проинтерпретировать результаты.
# Добавляем интеракцию
fit7 <- lm(scale(alc) ~
scale(agea) * gender + scale(eduyrs) +
scale(sclmeet) + icpart1 + icpdwrk + domicil,
data= ess7.RU
)
# Смотрим на результаты
summary(fit7)>
> Call:
> lm(formula = scale(alc) ~ scale(agea) * gender + scale(eduyrs) +
> scale(sclmeet) + icpart1 + icpdwrk + domicil, data = ess7.RU)
>
> Residuals:
> Min 1Q Median 3Q Max
> -1.9238 -0.5596 -0.2517 0.2858 6.0092
>
> Coefficients:
> Estimate Std. Error t value Pr(>|t|)
> (Intercept) 0.152166 0.139948 1.087 0.27720
> scale(agea) 0.292984 0.054334 5.392 8.96e-08 ***
> genderFemale -0.554140 0.068327 -8.110 1.71e-15 ***
> scale(eduyrs) -0.094178 0.036978 -2.547 0.01104 *
> scale(sclmeet) -0.007048 0.034738 -0.203 0.83926
> icpart1 0.106477 0.068168 1.562 0.11866
> icpdwrk -0.049712 0.076444 -0.650 0.51566
> domicil2 0.513659 0.193918 2.649 0.00822 **
> domicil3 0.115680 0.075038 1.542 0.12353
> domicil4 0.028053 0.085558 0.328 0.74308
> domicil5 0.154685 0.478511 0.323 0.74657
> scale(agea):genderFemale -0.360537 0.076133 -4.736 2.55e-06 ***
> ---
> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
>
> Residual standard error: 0.9481 on 871 degrees of freedom
> (1562 observations deleted due to missingness)
> Multiple R-squared: 0.1132, Adjusted R-squared: 0.102
> F-statistic: 10.11 on 11 and 871 DF, p-value: < 2.2e-16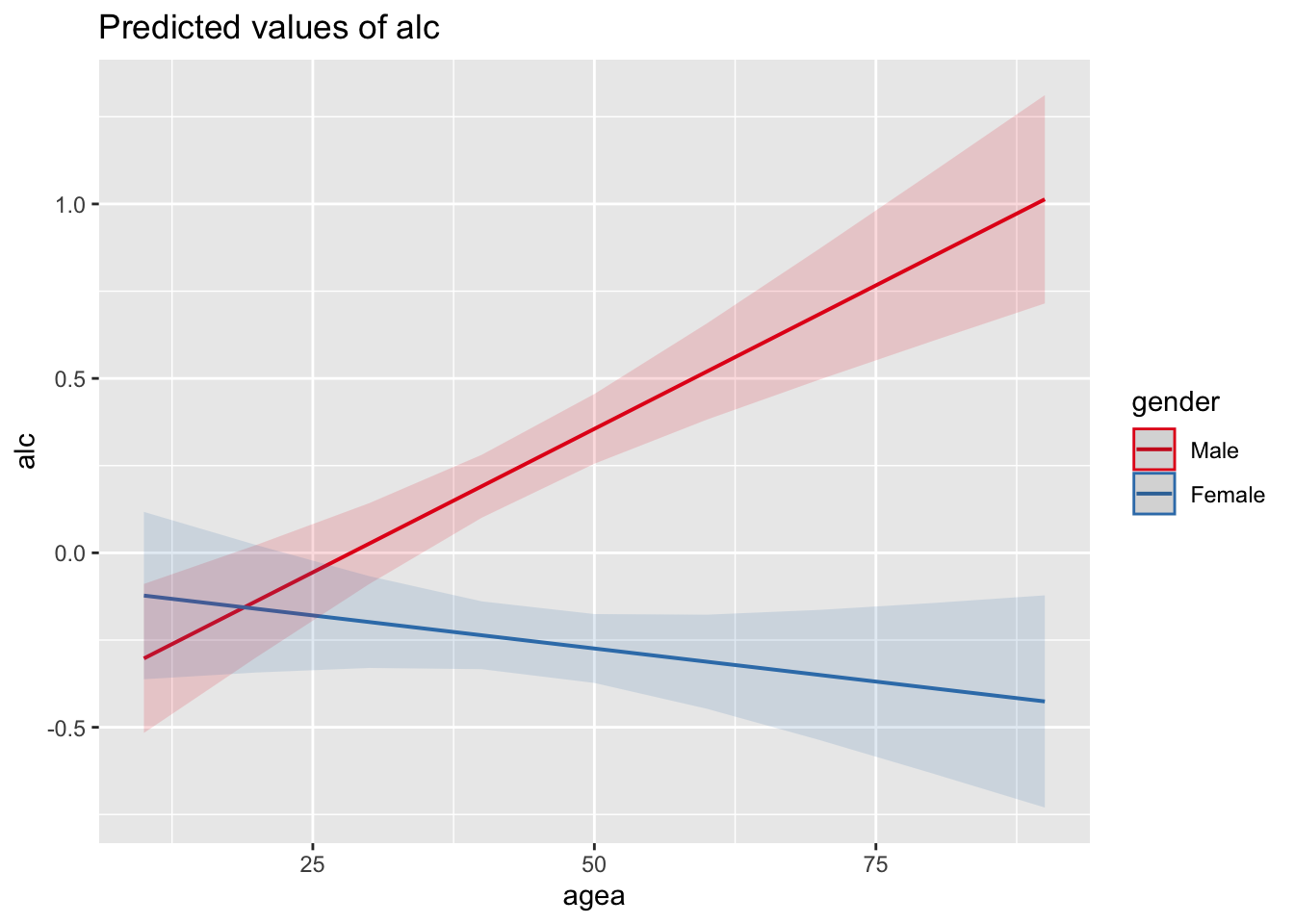
- Добавьте в модель квадратичный эффект возраста и посмотрите на то, как изменились результаты.
# Добавили квадрат возраста
fit8 <- lm(scale(alc) ~
scale(agea) * gender + scale(eduyrs) + I(agea^2) +
scale(sclmeet) + icpart1 + icpdwrk + domicil,
data= ess7.RU
)
# Смотрим результаты
summary(fit8)>
> Call:
> lm(formula = scale(alc) ~ scale(agea) * gender + scale(eduyrs) +
> I(agea^2) + scale(sclmeet) + icpart1 + icpdwrk + domicil,
> data = ess7.RU)
>
> Residuals:
> Min 1Q Median 3Q Max
> -1.6677 -0.5746 -0.2401 0.2911 6.0143
>
> Coefficients:
> Estimate Std. Error t value Pr(>|t|)
> (Intercept) 1.5409153 0.3333232 4.623 4.36e-06 ***
> scale(agea) 1.3850904 0.2444472 5.666 1.98e-08 ***
> genderFemale -0.5970626 0.0682043 -8.754 < 2e-16 ***
> scale(eduyrs) -0.1157746 0.0368642 -3.141 0.00174 **
> I(agea^2) -0.0006891 0.0001505 -4.580 5.34e-06 ***
> scale(sclmeet) 0.0078545 0.0345005 0.228 0.81996
> icpart1 0.1679374 0.0687230 2.444 0.01474 *
> icpdwrk 0.0902557 0.0815278 1.107 0.26858
> domicil2 0.4782636 0.1918879 2.492 0.01287 *
> domicil3 0.1213557 0.0742023 1.635 0.10231
> domicil4 0.0048130 0.0847454 0.057 0.95472
> domicil5 0.0675881 0.4734994 0.143 0.88653
> scale(agea):genderFemale -0.3309026 0.0755521 -4.380 1.33e-05 ***
> ---
> Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
>
> Residual standard error: 0.9374 on 870 degrees of freedom
> (1562 observations deleted due to missingness)
> Multiple R-squared: 0.1341, Adjusted R-squared: 0.1222
> F-statistic: 11.23 on 12 and 870 DF, p-value: < 2.2e-16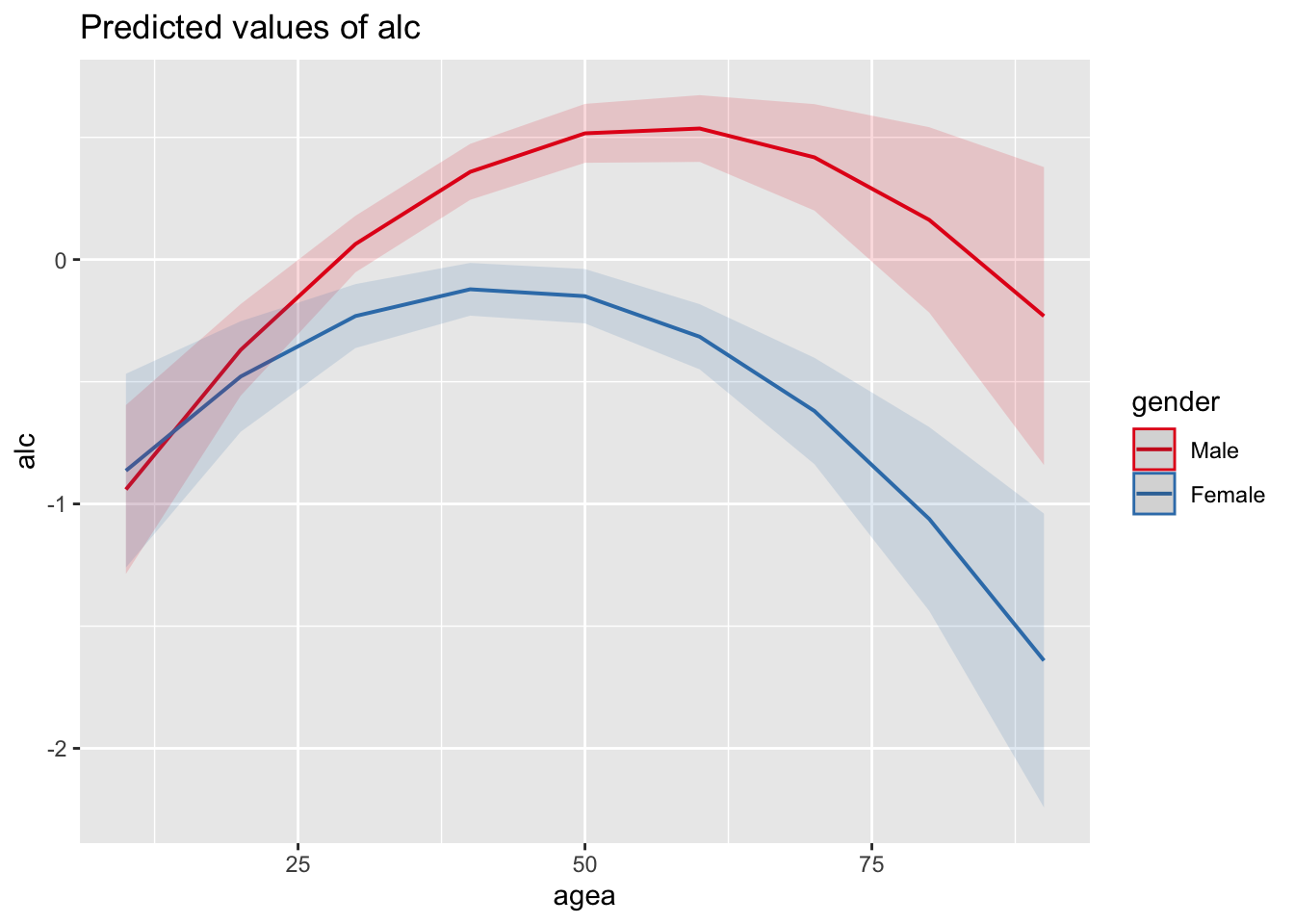
- Сравните полученные модели между собой: стабильность коэффициентов и стандартных ошибок, качество объяснения и нарушение допущений.
# Сводим результаты последних трех моделей в едтиную таблицу
regtab <- htmlreg(list(fit6, fit7, fit8))
browsable(HTML(regtab))| Model 1 | Model 2 | Model 3 | |
|---|---|---|---|
| (Intercept) | 0.15 | 0.15 | 1.54*** |
| (0.14) | (0.14) | (0.33) | |
| scale(agea) | 0.13** | 0.29*** | 1.39*** |
| (0.04) | (0.05) | (0.24) | |
| genderFemale | -0.50*** | -0.55*** | -0.60*** |
| (0.07) | (0.07) | (0.07) | |
| scale(eduyrs) | -0.09* | -0.09* | -0.12** |
| (0.04) | (0.04) | (0.04) | |
| scale(sclmeet) | -0.00 | -0.01 | 0.01 |
| (0.04) | (0.03) | (0.03) | |
| icpart1 | 0.06 | 0.11 | 0.17* |
| (0.07) | (0.07) | (0.07) | |
| icpdwrk | -0.04 | -0.05 | 0.09 |
| (0.08) | (0.08) | (0.08) | |
| domicil2 | 0.56** | 0.51** | 0.48* |
| (0.20) | (0.19) | (0.19) | |
| domicil3 | 0.14 | 0.12 | 0.12 |
| (0.08) | (0.08) | (0.07) | |
| domicil4 | 0.03 | 0.03 | 0.00 |
| (0.09) | (0.09) | (0.08) | |
| domicil5 | 0.05 | 0.15 | 0.07 |
| (0.48) | (0.48) | (0.47) | |
| scale(agea):genderFemale | -0.36*** | -0.33*** | |
| (0.08) | (0.08) | ||
| agea^2 | -0.00*** | ||
| (0.00) | |||
| R2 | 0.09 | 0.11 | 0.13 |
| Adj. R2 | 0.08 | 0.10 | 0.12 |
| Num. obs. | 883 | 883 | 883 |
| ***p < 0.001; **p < 0.01; *p < 0.05 | |||
Данные: Datafile_ESS7.sav
По итогам попробуйте составить целиковый работающий скрипт. Очистите память, перезапустите сессию R и запустите весь скрипт целиком. Если скрипт не запускается, найдите в нем ошибки и доведите код до безошибочного вида.
